[ChEMBL compounds->Target->gene->disease relationship]に加えて[CheEMBL Interaction->Assay->publication->MeSH(disease term)]を使用します。但し,同じ出版物に掲載されていても,化合物と疾患の間に相関があるという保証はありません。
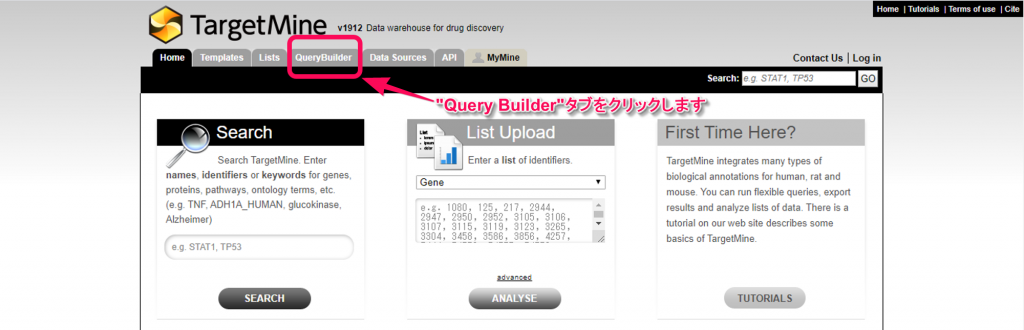
1.TargetMineのメインページで,”Query Builder”タブをクリックします。
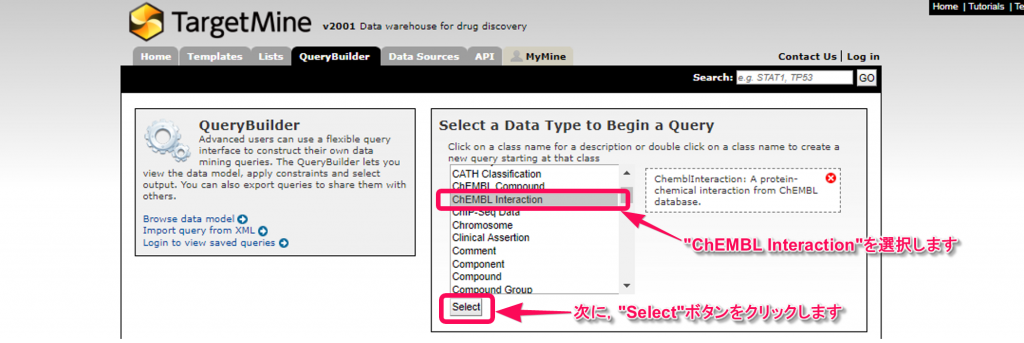
2. “ChEMBL Interaction”を選択した後,”Select”ボタンをクリックして,クエリを開始します。
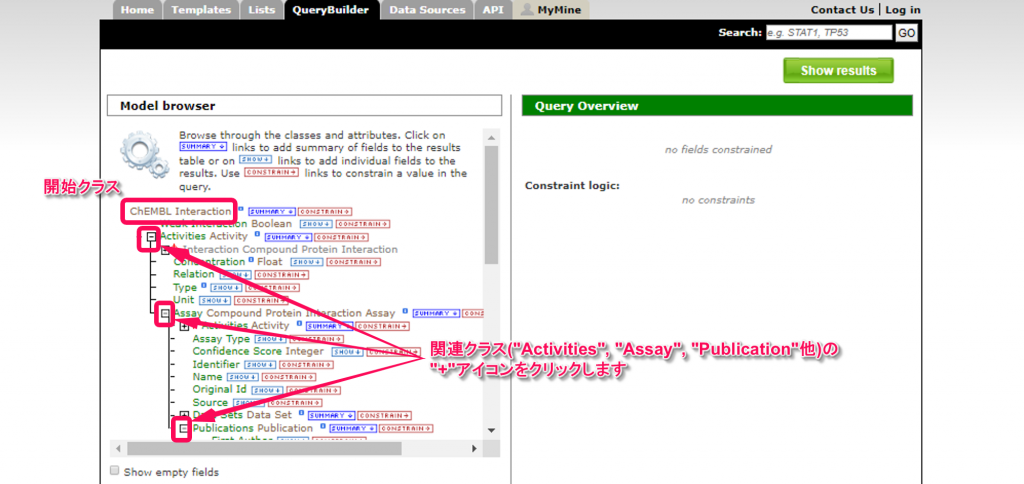
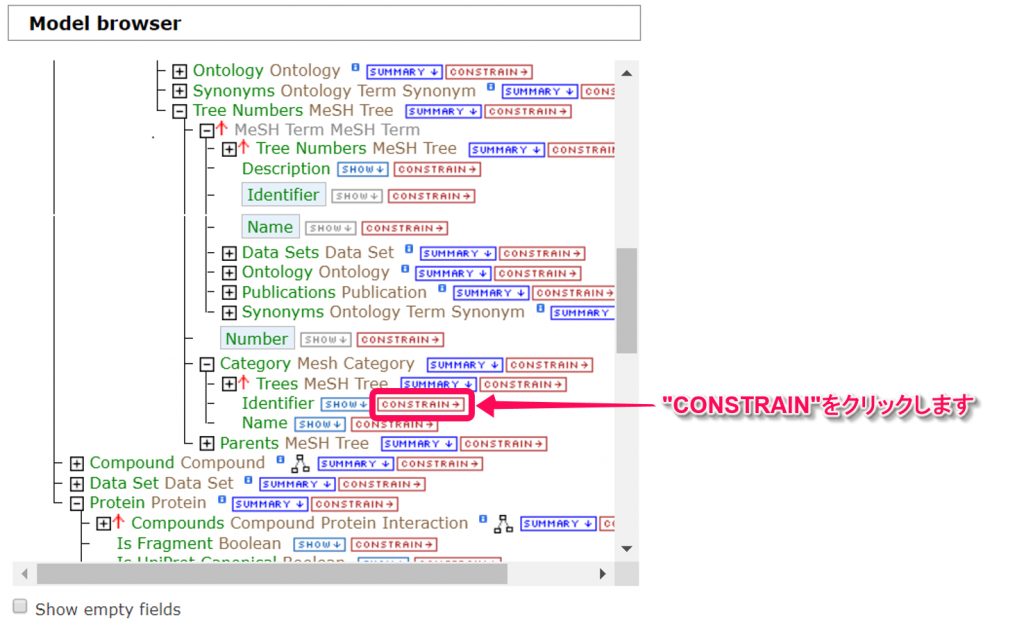
3. “Model browser”でTargetMineのデータモデルを表示させることができます。データモデルは,ツリー構造をしており,”+”をクリックすることで,サブクラスが表示できます。例えば,”ChEMBL Interaction”クラスをルートとして,サブクラスの”Activities”をクリックすると,さらにそのサブクラスの”Assay”が現れます。”ChEMBL Interaction”からはじめて,”Category”を表示させるには,”Activities”,”Assay”,”Publications”,”Mesh Terms”,”Tree Numbers”,”Category”と順に展開します。同じく”MeSH Term”(“ChEMBL Interaction”->”Activities->”Assay”->”Publications”->”Mesh Terms->”Tree Numbers->”Parents”->”MeSH Term”),”Compound”(“ChEMBL Interaction”->”Compound”),”Organism”(“ChEMBL Interaction”->”Protein”->”Organism”)も展開して表示できます。
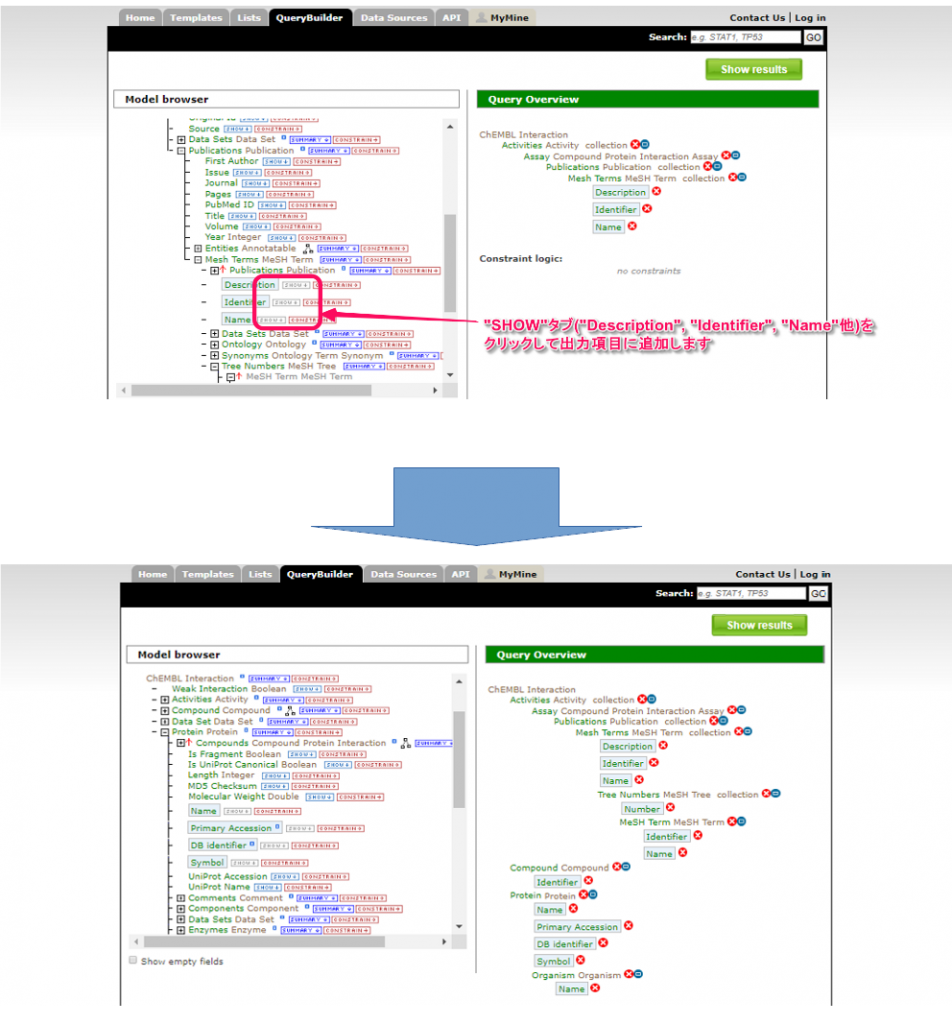
4. 次に各々のクラスについて,属性の”SHOW”タブをクリックすることで出力テーブルに追加することができます。例えば,”Mesh Terms”クラスの”Description”,”Identifier”,”Name”。”Tree Numbers”クラスの”Number”。”MeSH Term”クラスの”Identifier”,”Name”。”Compound”クラスの”Identifier”。”Protein”クラスの”Name”,”Primary Accession”,”DB identifier”,”Symbol”。”Organism”クラスの”Name”を選択します。
選んだ項目は”Query Overview”に表示されます。
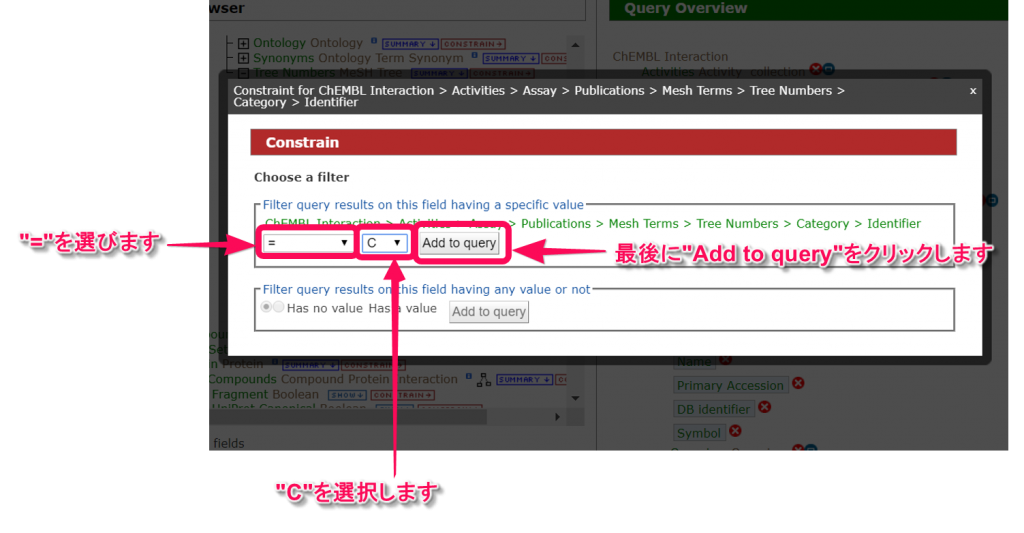
5. このチュートリアルでは,結果出力をすぐに得られるようにMesh Categoryに制限を付けます。”Category”クラス配下の”Identifier”クラスの’CONSTRAIN’タブをクリックし,ポップアップ画面で,プルダウンリストから”C”を選択し”Add to query”ボタンをクリックします。
制限なしの検索はサーバに負荷がかかりますので,クエリーをサブミットする前に(お問い合わせへのリンク)ご相談ください。
尚,制限なしの場合の結果はこちらからダウンロードできます。
6. ”Fields selected for output”エリアで,出力カラムに対応する項目のボックスをドラッグ&ドロップすることで,結果テーブルのカラムの並びを変更できます。
(ケース1.1の手順6を参照ください)
7. “Show results”ボタンをクリックして結果テーブルを表示します。
(ケース1.1の手順7を参照ください)
クエリーの結果の確認と保存については,ケース1.1の手順8,9を参照ください。