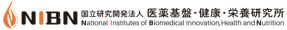TargetMineは,以下の研究でデータ分析に利用されています。
- HCV pathogenesis (yeast two-hybrid assays): Tripathi et al. 2010.
- HCV pathogenesis (proteomic data): Tripathi et al. 2012.
- Lung tumorigenesis (gene expression): Ihara et al. 2012.
- HCV pathogenesis (yeast two-hybrid assays and literature): Tripathi et al. 2013.
- Cancer immunotherapy (single-cell gene-expression profiles): Nakae, et al. 2015.
- Alzheimer’s disease (genome-wide siRNA screening): Camargo, et al. 2015.
- Colitis (microRNA): Lee et al. 2016.
- Idiopathic interstitial pneumonias (proteomic data): Hamano et al. 2017.
- COPD and IPF (gene expression): Tripathi et al. 2020.
上記研究のいくつかについて,概要を以下に示します。
応用例1
詳細は図をクリックして下さい。
56のたんぱく質が酵母2ハイブリッドスクリーニングで識別されました。タンパク質を相互作用するパートナーと組み合わせて,拡張ネットワークを構築しました。優先順位づけされた4つのターゲットは,エノラーゼ(ENO1),パキシリン(PXN),SLC25A5,及びビンキュリン(VCL)です。
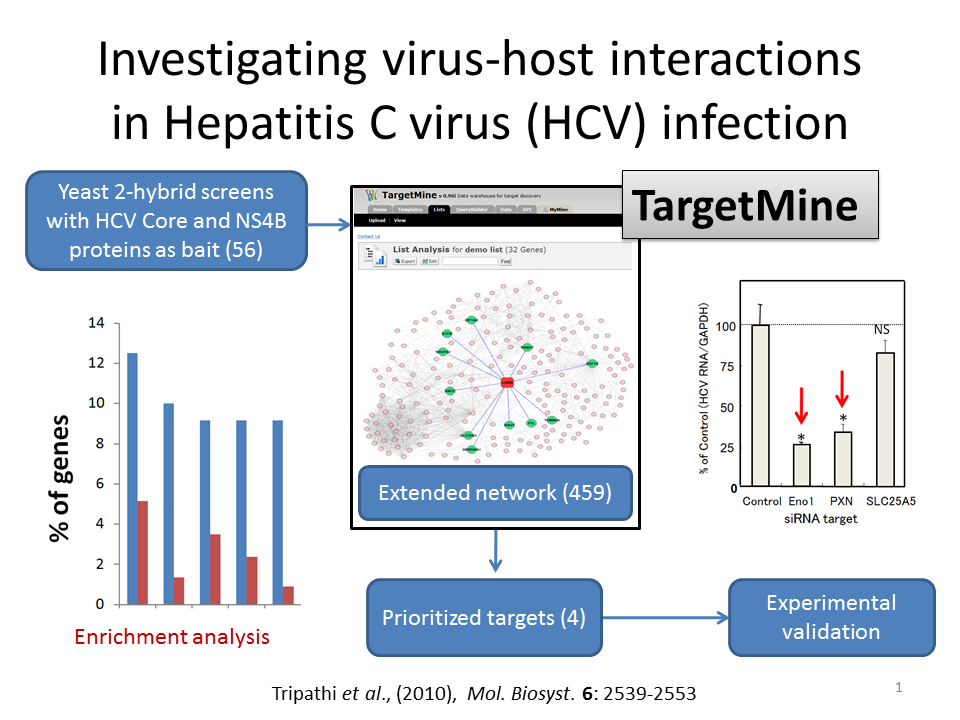
応用例2
詳細は図をクリックして下さい。
HCV感染におけるPA28-γの役割を理解するために,肝臓(HCV病因のin vivoモデル)でHCVコアタンパク質を発現するトランスジェニックマウスの細胞タンパク質レベルを,PA28-γ発言のノックアウト有無にかかわらず,比較,及び調査しました。
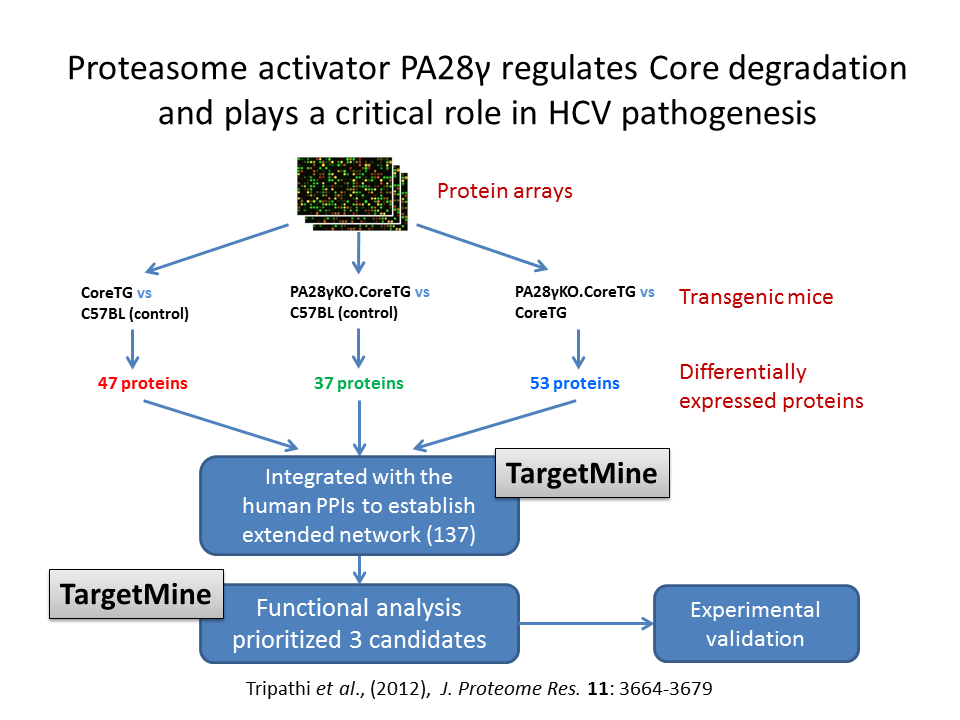
応用例3
詳細は図をクリックして下さい。
単一細胞遺伝子発現プロファイルの83の遺伝子は,主成分分析(PCA)によって2つの異なるグループに分けられました。各グループにおいて単一細胞の含有物に積極的に寄与する遺伝子は,PC1ローディング値によって特定され,遺伝子オントロジー(GO)エンリッチメント分析のテーマを実行しました。この二つのグループは,ダイバーズGOの条件についてエンリッチされていることが分かり,”アクティブ化”と”無活動”という2つの異なる状態の存在を明らかにしました。
参考文献:
- Tripathi LP, Itoh MN, Takeda Y, Tsujino K, Kondo Y, Kumanogoh A and Mizuguchi K (2020) Integrative Analysis Reveals Common and Unique Roles of Tetraspanins in Fibrosis and Emphysema. Front. Genet. 11:585998. [doi: 10.3389/fgene.2020.585998]
- Hamano Y, et al. (2017) Classification of idiopathic interstitial pneumonias using anti–myxovirus resistance-protein 1 autoantibody. Sci Rep 7, 43201. [https://doi.org/10.1038/srep43201]
- Lee J, et al. (2016) Profiles of microRNA networks in intestinal epithelial cells in a mouse model of colitis. Sci Rep 5, 18174. [https://doi.org/10.1038/srep18174]
- Nakae Y, et al. (2015) Two distinct effector memory cell populations of WT1 (Wilms’ tumor gene 1)-specific cytotoxic T lymphocytes in acute myeloid leukemia patients. Cancer Immunol Immunother. 64(7):791-804. [PubMed:25835542]
- Camargo LM, et al. (2015) Pathway-based analysis of genome-wide siRNA screens reveals the regulatory landscape of APP processing. Plos ONE 10(2):e0115369. [PubMed:25723573]
- Tripathi LP, et al. (2013) Understanding the Biological Context of NS5A-Host Interactions in HCV Infection: A Network-Based Approach. J Proteome Res. [PubMed:23682656]
- Ihara S, et al. (2012) Inhibitory Roles of Signal Transducer and Activator of Transcription 3 in Antitumor Immunity during Carcinogen-Induced Lung Tumorigenesis. Cancer Res. 72(12):2990-2999. [PubMed:22659452]
- Tripathi LP, et al. (2012) Proteomic Analysis of Hepatitis C Virus (HCV) Core Protein Transfection and Host Regulator PA28γ Knockout in HCV Pathogenesis: A Network-Based Study. J Proteome Res. 11: 3664-3679. [PubMed:22646850]
- Tripathi LP, et al. (2010) Network based analysis of hepatitis C virus Core and NS4B protein interactions. Mol Biosyst 6: 2539-2553. [PubMed:20953506 ]