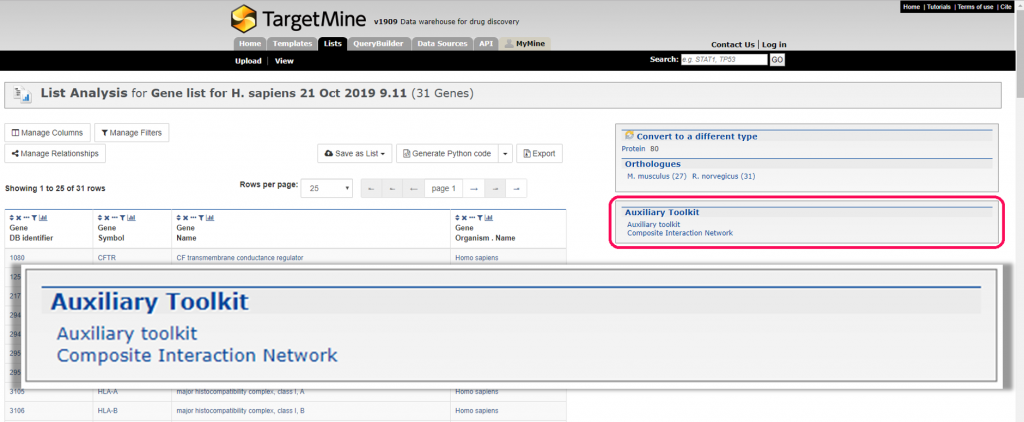
 TargetMineの補助ツールキットは、TargetMinデータウェアハウスに組み込まれた生物学的なデータを検索し解析するためのインタラクティブなデータ解析機能を特徴としています。TargetMineユーザーは、このツールキットを使うことで、複雑な検索をインタラクティブに実行し、理解しやすい出力形式での検索結果を得ることで、新しい仮説の発見をすることができます。補助ツールキットは現在のところ、TargetMineを右の2つの方法で補強しています: 1) 自動的なデータ変換とグラフ、チャートのついた生物学的なエンリッチメント解析。 2) 合成相互作用ネットワーク。これらの機能には、TargetMineのリストページか、(以下に示す)専用の入力フォームから利用できます。
TargetMineの補助ツールキットは、TargetMinデータウェアハウスに組み込まれた生物学的なデータを検索し解析するためのインタラクティブなデータ解析機能を特徴としています。TargetMineユーザーは、このツールキットを使うことで、複雑な検索をインタラクティブに実行し、理解しやすい出力形式での検索結果を得ることで、新しい仮説の発見をすることができます。補助ツールキットは現在のところ、TargetMineを右の2つの方法で補強しています: 1) 自動的なデータ変換とグラフ、チャートのついた生物学的なエンリッチメント解析。 2) 合成相互作用ネットワーク。これらの機能には、TargetMineのリストページか、(以下に示す)専用の入力フォームから利用できます。
生物学的なエンリッチメント解析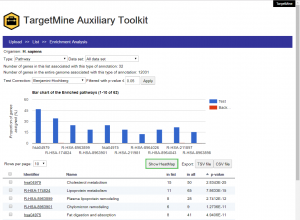
ユーザーは遺伝子のリストをアップロードし、マウスのわずか数クリックで、以降のエンリッチメント解析のために、PPIの相互作用相手を含むようにクエリーを拡張し、オーソログ変換をすることができます。「Show HeatMap」タブをクリックすることで、ユーザーは遺伝子と生物学的テーマのアノテーションをヒートマップで見ることができます。エンリッチメント解析の結果は、バックグラウンドに対してエンリッチされた生物学的テーマの分布の表とヒストグラムの形式で得ることができます(ホールゲノムアノテーション)。出力の表(とヒストグラム)を、P値や注釈された遺伝子(フォアグラウンド、バックグラウンド)数、生物学的テーマの名称などによりソートすることができます。KEGGパスウェイのエンリッチメント解析の場合は、出力表のパスウェイ名のところをクリックすることで、関連する遺伝子がKEGGのウェブサイトへ送られ、対応するパスウェイ上でそれらがハイライトされます。
合成相互作用ネットワーク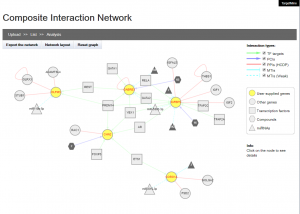
遺伝子のリストを入力することで、それらと関連づいたTargetMine中の全ての生体分子相互作用(PPI、MTI、PCI/医薬品ターゲット相互作用、転写因子とターゲット遺伝子の相互作用など)を合成した相互作用ネットワークを構築し可視化することができます。合成された相互作用ネットワークの上で、生体分子相互作用は異なる種類ごとに異なるシンボルと色で表現され、ユーザーはレイアウトの描画方法を、力学モデル、環状モデル、有機モデルなどから選択でき、特定の相互作用種類のものの表示/非表示を切り替えることができます。PCI(ChEMBLのみ)を、望ましい活性値を閾値として絞り込んで表示することもできます。
[Composite Interaction Network]
エンリッチメント解析ウェブサービス
生物学的テーマのエンリッチメント解析ウェブサービス。アカウントやログインは不要です。