‘Gene Disease Pair’は,様々なソースによって遺伝子と疾患のオントロジー用語が関連付くように索引付けします。
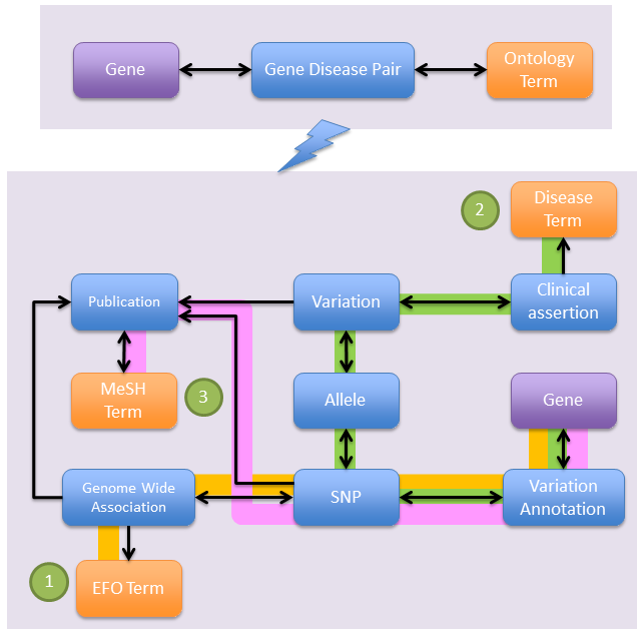
遺伝子はEFO①,MedGen②,及びMeSH③の疾患のオントロジー用語に,各々GWAS(朱色ライン),ClinVar(緑色ライン),及びdbSNP(桃色ライン)のSNPデータを介して関連付けされます。
Ex. ClinVarベースの化合物-疾患データセットの作成
ケース1.2ではClinVar,GWAS,及びdbSNP全てで疾患を関連付けていますが,”Disease Term”に制限を追加することで,いずれかのソースによってのみ関連付けることもできます。
例えば,ClinVarのみによって関連付けした化合物-疾患データセットを作成する手順は次のようになります。
ケース1.2の手順5の後に,”Disease Term”クラスの”CONSTRAIN”タブをクリックし,”Constrain”ポップアップ画面で,見出し’Filter query results based on this field being a member of a specific class of objects’をクリックして,”Constrained to be in subclass”のプルダウンメニューから”Disease Term”を選択し,最後に’Add to query’ボタンをクリックします。
![疾患用語のサブクラスと限定するソースの対応関係 [サブクラス] - [ソース] 'Disease Term' – 'ClinVar' 'EOF Term' - 'GWAS' 'MeSH Term' - 'dbSNP'](https://targetmine.mizuguchilab.org/wp51/wp-content/uploads/2020/03/GeneDiseasePairClass-8-14_jp-2-724x1024.png)
疾患用語のサブクラスと限定するソースの対応関係
[サブクラス] – [ソース]
‘Disease Term’ – ‘ClinVar’
‘EOF Term’ – ‘GWAS’
‘MeSH Term’ – ‘dbSNP’