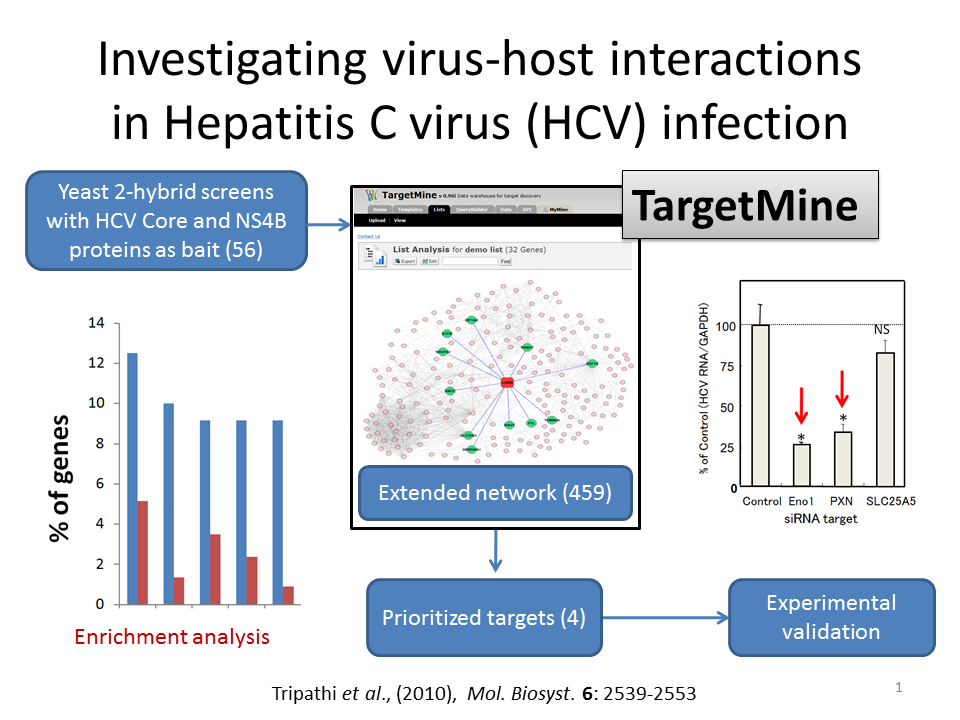
 HCVの病因におけるHCVと宿主の相互作用を理解するための,一連の酵母2ハイブリッド(Y2H)スクリーンの使用は,CoreとNS4B(Coreの11個の相互作用物質とNS4Bの45個の相互作用物質)と相互作用する56のヒト宿主タンパク質の同定につながりました。TargetMineの助けを借りて拡張PPIネットワークを推測し,HCV感染におけるコアとNS4Bの相互作用の生物学的な意味をさらに理解するために,このY2H相互作用を,ヒトのインタラクトームと統合しました。次に,拡張PPIネットワークを,特定の生物学関連(KEGG経パスウェイ,GO生物学的プロセス,OMIM表現型)のエンリッチメントについて調査しました。エンリッチされた生物学的テーマと拡張ネットワークの関連遺伝子の観察により,459の候補のうち4つがHCV複製と宿主の存続の推定標的であることが示唆されました。
HCVの病因におけるHCVと宿主の相互作用を理解するための,一連の酵母2ハイブリッド(Y2H)スクリーンの使用は,CoreとNS4B(Coreの11個の相互作用物質とNS4Bの45個の相互作用物質)と相互作用する56のヒト宿主タンパク質の同定につながりました。TargetMineの助けを借りて拡張PPIネットワークを推測し,HCV感染におけるコアとNS4Bの相互作用の生物学的な意味をさらに理解するために,このY2H相互作用を,ヒトのインタラクトームと統合しました。次に,拡張PPIネットワークを,特定の生物学関連(KEGG経パスウェイ,GO生物学的プロセス,OMIM表現型)のエンリッチメントについて調査しました。エンリッチされた生物学的テーマと拡張ネットワークの関連遺伝子の観察により,459の候補のうち4つがHCV複製と宿主の存続の推定標的であることが示唆されました。
4つのターゲット,エノラーゼ(ENO1),パキシリン(PXN),SLC25A5,およびビンキュリン(VCL)を,siRNAを使用した細胞アッセイにより,HCVの複製および放出について起こりうる機能について評価しました。ENO1 siRNAノックダウンは,HCV遺伝子型1bおよび2aのHCV複製,及び遺伝子型2aのウイルスRNA放出を阻害することが分かりました。PXN siRNA阻害が,遺伝子型1bで,特異的に複製を阻害することが観察されましたが,遺伝子型2aではそうではありませんでした。一方,SLC25A5 siRNAは,遺伝子型2aのウイルスRNA放出のわずかな増加を促進しました。これらの結果は,優先順位付けされた4遺伝子のうち少なくとも2つが,HCV感染を抑制するための新規の潜在薬物標的であることを示唆しています。
参考文献:
- Tripathi LP, et al. (2010) Network based analysis of hepatitis C virus Core and NS4B protein interactions. Mol Biosyst 6: 2539-2553. [PubMed:20953506 ]