TargetMineのリスト機能を使うと、ユーザーが入力したオブジェクトのリスト(例:遺伝子やタンパク質のリスト)を使ったクエリーを実行できます。ユーザーは、検索結果ページを元にリストを作ったり、IDのリストをアップロードすることで作ることもできます。リストは、テンプレートクエリーやクエリービルダーでクエリーの制約条件として用いることもできますし、またリストの論理演算(和、積、対称差)をして結果をエキスポートすることもできます。ユーザーは、リストを検索したりコピーしたりすることもできます。
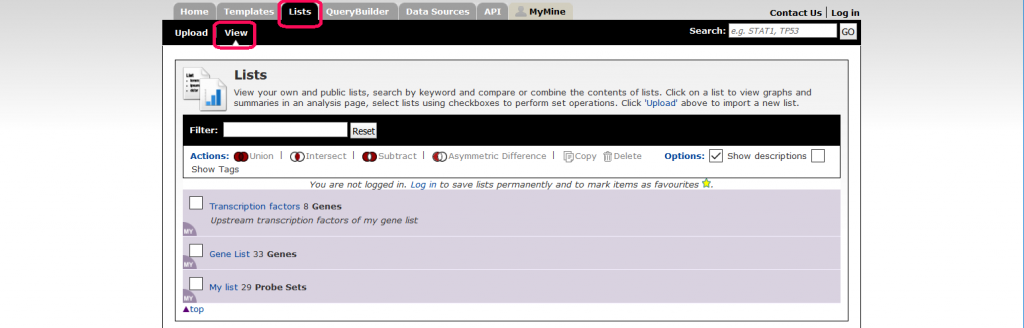
- リストを見る
リストページは、TargetMineのメニューバーの「Lists」から見ることができます。
- リストをアップロードする
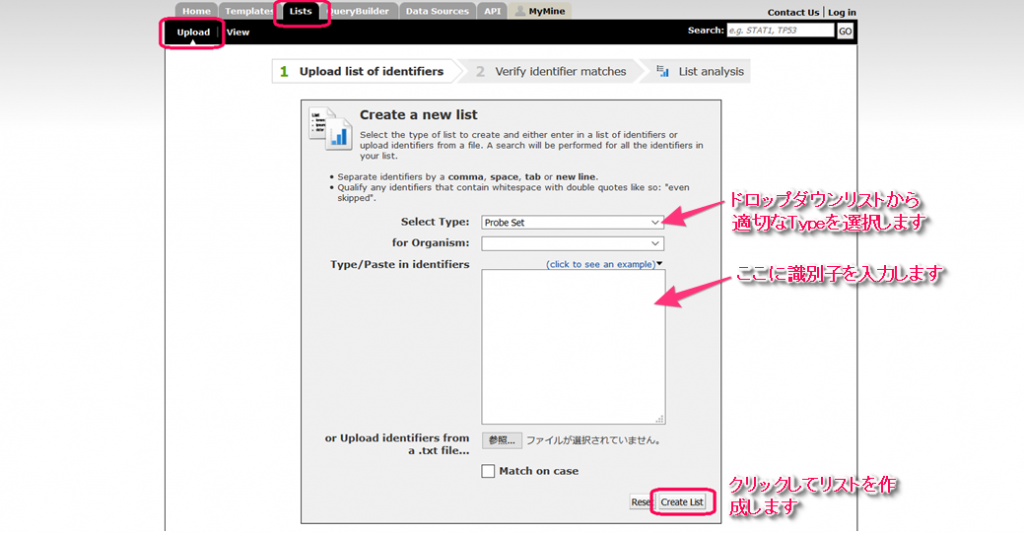
ユーザーは、リストページの「Upload」タブで、「Type/Paste in identifiers」で識別子を入力するか、オブジェクトのリストを含むファイルをアップロードすることによりリストを作ります。
リストは、オブジェクトのただ一種類の「type」(例: 遺伝子、タンパク質)から作成され、それはドロップダウンリストで設定できます。しかし、リストは複数の識別子の種類(例: 遺伝子シンボルと遺伝子のID)を含むことも可能です。必要に応じて、識別子のリストに対して生物種を設定することもできます。
- リストの確認
リスト作成前に、TargetMineのデータベースを用いたチェックがされ、不一致のものは‘No matches found’に出力されます。リストに名前をつけるか、自動生成されたデフォルトの名前を用います。
- リスト解析ページ
‘List Analysis’ページでは、リストの中身の詳細に加えて、付加情報を見ることができます。リストが作成されると、解析ページが自動的に表示されます。いつでも、‘List’タブの‘View’を訪問することで閲覧することができます。
-
- リストの中身の概要は、リストのオブジェクトの表、リストの解説、リストの情報、(もしある場合は)変換ツールや外部リンク、を含みます。
- ‘widgets’のシリーズは、大きな比率を占めるKEGGパスウェイ、Gene Ontology (GO)ターム、化合物、Disease Ontologyとの関連性、CATHの分類など、リストに関する追加情報を表示します。
- リストの中の一群のオブジェクトを使ってあらかじめ実行されたテンプレートクエリーのセット